Todos tenemos en casa rincones oscuros donde se agazapa alguna regleta con infinidad de cables enchufados, y no sabemos cómo acaban anudados. ¿Cómo es posible que cada una de nuestras células contenga más de dos metros de ADN que continuamente se está enrollando y desenrollando, abriendo y cerrando, e incluso copiándose en cadenas idénticas, sin que todo ello termine en un enredo imposible? El ADN tiene su propia matemática, y al sistema que lo controla no solo le debemos la vida, sino también herramientas que nos ayudan a luchar contra enfermedades.

Una vez que Watson, Crick, Franklin, Wilkins y otros desvelaron la estructura básica del ADN como una doble hélice, comenzó a descubrirse que la organización de esta molécula en las células era un prodigio de orden: la doble hélice forma vueltas alrededor de minúsculos carretes compuestos por ocho proteínas llamadas histonas. Cada carrete de histonas con su ADN alrededor es un nucleosoma; unidos entre sí por el hilo de ADN ofrecen el aspecto de un collar de cuentas, una estructura fotografiada por primera vez al microscopio electrónico en 1974 por Ada y Donald Olins. Pero este collar de nucleosomas a su vez se enrolla en una hélice, y esta en otra más gruesa, hasta formar una miríada de lazos superenrollados y compactados en torno a un andamio de proteínas; un cromosoma.
La topología del ADN
Una célula humana contiene 23 pares de cromosomas, cada uno de ellos formado por una sola cadena doble de ADN superenrollada y compactada con diversas proteínas. Si estiráramos todo el ADN de los 46 cromosomas obtendríamos esa longitud de dos metros. Pero si hiciéramos lo mismo con el ADN de todas las células de un cuerpo humano, se ha estimado que el resultado sería una doble hebra de más de 100.000 millones de kilómetros.

Los libros de texto representan los cromosomas como estructuras en forma de X, pero esto solo se observa cuando la célula va a dividirse. Cada uno de los dos brazos de la X es una cromátida; ambas son copias idénticas que se repartirán entre las dos células hijas. Sin embargo, para llegar a ese momento ha sido necesario que todo el ADN se duplique, y para ello debe desenrollarse, ya que cuando está compactado es inutilizable. Cuando la célula no se está dividiendo —en interfase—, el ADN está desenrollado en una masa difusa, la cromatina, compuesta por collares de nucleosomas; pero donde cada cromosoma conserva su territorio en el núcleo celular, como plastilinas de colores formando una pelota sin que se mezclen.

Es aquí donde entra en juego la topología, la rama de las matemáticas que estudia las propiedades de los cuerpos geométricos cuando se doblan, estiran, retuercen o aplastan manteniendo su integridad estructural, como un aro de goma elástica que puede adoptar distintas formas. La topología del ADN todavía es una ciencia en construcción. “Ha sido un misterio cómo los cromosomas descondensados en la interfase permanecen esencialmente sin nudos”, escribía un grupo de investigadores en un estudio de 2018 que ilustra cómo los científicos utilizan complejas herramientas matemáticas de simulación y teoría de nudos para entender los mecanismos que mantienen el orden en esa maraña sin que el ADN se convierta en un amasijo.
Modelado molecular que salva vidas
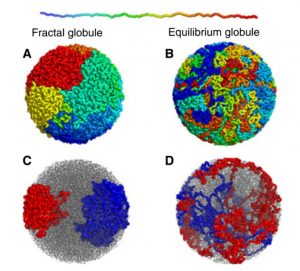
Según la teoría actual, los cromosomas desenrollados en el núcleo de la célula forman glóbulos fractales. A diferencia del glóbulo en equilibrio, que sería la maraña embrollada de las luces del árbol de Navidad, un glóbulo fractal es una estructura compactada al máximo, pero ordenada de modo que se mantiene un camino hamiltoniano; así llamado cuando se puede seguir el hilo pasando por todos los puntos, sin pasar dos veces por el mismo y de modo que los caminos no se cortan, por lo que no hay nudos. Es un ejemplo de la curva de Peano, un patrón que puede llenar todo un plano con una forma que se repite pero que sigue un camino lineal sin cruzamientos. Y que adopta un patrón cuya estructura se itera desde pequeña a gran escala, como ocurre en la geometría fractal.
Naturalmente, esta estructura ordenada no se mantiene sola, sino gracias a una maquinaria celular que deshace los nudos y entrecruzamientos, como los que se producen cuando un anillo circular de ADN —como los cromosomas de las bacterias— se replica, dando lugar a dos aros entrelazados. Las enzimas topoisomerasas cambian la topología del ADN para resolver estos problemas: las de tipo I cortan y reconectan solo una de las hebras de la doble cadena de ADN, mientras que las de tipo II lo hacen con las dos cadenas. Por otra parte, en la compactación de los cromosomas para la división celular intervienen otras enzimas como la cohesina, una proteína con forma de esposas que sirve para condensar los lazos de ADN y mantener las cromátidas juntas, pero no revueltas.
Las topoisomerasas requieren una regulación muy fina: si no funcionan, la célula muere, pero también si hay un exceso de cortes que fragmentan el ADN irremediablemente. Es por ello que estas enzimas son el objetivo de fármacos: antibióticos (quinolonas como el ciprofloxacino) frente a infecciones bacterianas y quimioterapias contra el cáncer. Muchos de estos medicamentos no actúan inhibiendo las topoisomerasas, sino dejándolas actuar y estabilizando las roturas en el ADN para que la célula muera. Algunas quimioterapias contra el cáncer derivan de un inhibidor natural de topoisomerasas llamado camptotecina que se extrae de un árbol de China. Hoy el modelado molecular permite diseñar nuevas quimioterapias específicas dirigidas a las topoisomerasas: matemáticas que pueden salvar vidas.
Comentarios sobre esta publicación